在 rpart model 中大概有幾個比較重要的參數:
- weights: 用來給與data的weight,如果想加重某些data的權重時可使用。 (例如:Adaboost.M1 的演算法)
- method:分成 “anova”、”poisson”、”class”和”exp”。
- parms:splitting function的參數,會根據上面不同的方法給不同的參數。(例如:”anova”方法是不需要參數的)
- control: rpart.control object
以上是rpart的參數部分,大部份都是集中在選擇model和data的權重上。一旦決定方法後,在model中的重要參數,大部分都是在control中用一個rpart.control的物件進行設定的。
接下來我們來介紹 rpart.control中的幾個重要參數:
- minsplit:每一個node最少要幾個data
- minbucket:在末端的node上最少要幾個data
- cp:complexity parameter. (決定精度的參數)
- maxdepth:Tree的深度
rpart.control中的參數很多,半筆者比較常用的大概是上面幾個。
(附帶一提,rpart.control的參數,也可以直接掛在rpart的最後面”…”的地方。)
~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~
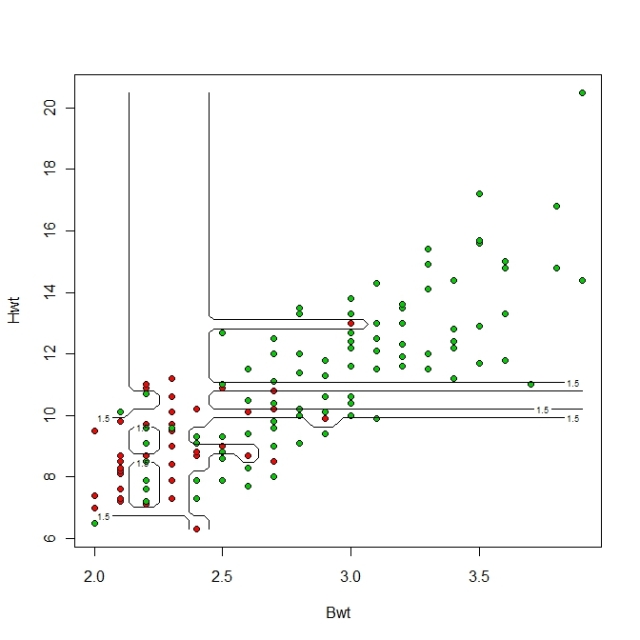
接下來,我們將使用cats data調整不同的cp和minsplit:
將前文中的這行
cats_rpart_model <- rpart(Sex~., data = cats)
改成這行
cats_rpart_model <- rpart(Sex~., data = cats, minsplit=1, cp=1e-3)
我們可以比較一下兩次的結果:
很明顯看得出來,右邊雖然誤差可能比較小,但應該是已經overfit了。
~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~
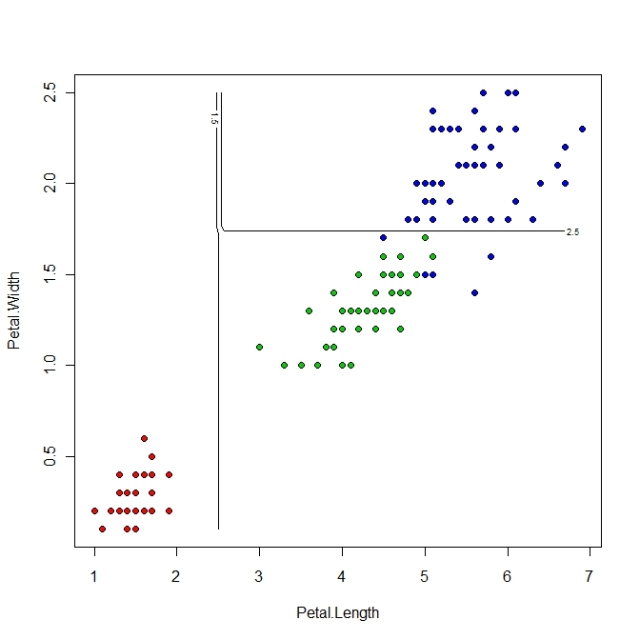
接下來我們測試iris data,只採用iris中的Petal.Length和Petal.Length兩個維度的Data。
rm(list=ls(all=TRUE))
data(iris)
Iris_2D <- iris[,3:5]
plot(Iris_2D[1:2], pch = 21, bg = c("red", "green3", "blue")[unclass(Iris_2D$Species)])
library(rpart)
Iris_2D_rpart_model <- rpart(Species~., data = Iris_2D)
Iris_2D_rpart_pred <- predict(Iris_2D_rpart_model, Iris_2D)
Iris_2D_rpart_pred_ClassN <- apply( Iris_2D_rpart_pred,1,function(one_row) return(which(one_row == max(one_row))))
Iris_2D_rpart_pred_Class <- apply( Iris_2D_rpart_pred,1,function(one_row) return(colnames(Iris_2D_rpart_pred)[which(one_row == max(one_row))]))
Iris_2D_Class_temp <- unclass(Iris_2D$Species)
Iris_2D_Class <- attr(Iris_2D_Class_temp ,"levels")[Iris_2D_Class_temp]
table(Iris_2D_rpart_pred_Class,Iris_2D_Class)
plot(Iris_2D_rpart_model)
text(Iris_2D_rpart_model)
x1 <- seq(min(Iris_2D$Petal.Length), max(Iris_2D$Petal.Length), length = 50)
x2 <- seq(min(Iris_2D$Petal.Width), max(Iris_2D$Petal.Width), length = 50)
Feature_x1_to_x2 <- expand.grid(Petal.Length = x1, Petal.Width = x2)
Feature_x1_to_x2_Class <- apply(predict(Iris_2D_rpart_model,Feature_x1_to_x2),1,
function(one_row) return(which(one_row == max(one_row))))
plot(Iris_2D[1:2], pch = 21, bg = c("red", "green3", "blue")[unclass(Iris_2D$Species)])
contour(x1,x2,matrix(Feature_x1_to_x2_Class,length(x1)),add = T, levels = c(1.5,2.5),labex = 0)
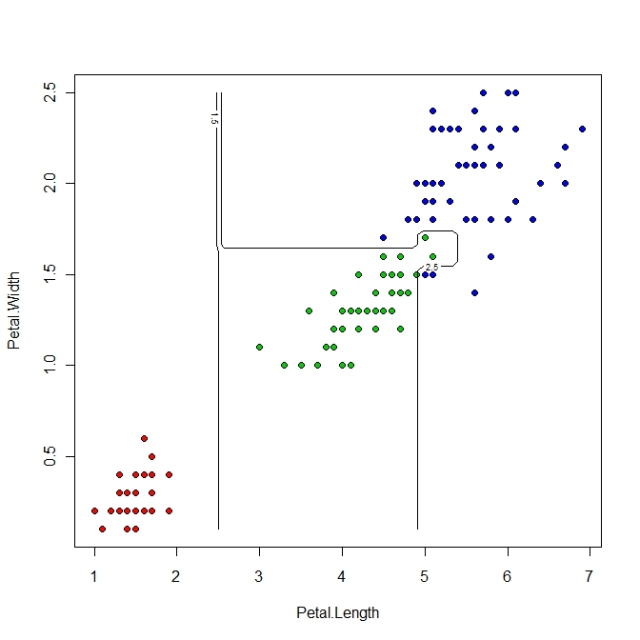
同樣的,我們可以把第7行的model換成下面這行在run一次
Iris_2D_rpart_model <- rpart(Species~., data = Iris_2D, minsplit=1, cp=1e-3)
我們會得到:
~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~
最後,在rpart中也有類似像svm中那個tune的設計。可以直接在圖上畫出各種cp所train出來的model的精度。如下:(延續使用上方的iris作為例子)
plotcp(Iris_2D_rpart_model) printcp(Iris_2D_rpart_model)
會得到cp和error的一張圖,如下
並且會在R的shell中看到以下的訊息:
> printcp(Iris_2D_rpart_model)
Classification tree:
rpart(formula = Species ~ ., data = Iris_2D, minsplit = 1, cp = 0.001)
Variables actually used in tree construction:
[1] Petal.Length Petal.Width
Root node error: 100/150 = 0.66667
n= 150
CP nsplit rel error xerror xstd
1 0.500 0 1.00 1.16 0.051277
2 0.440 1 0.50 0.65 0.060690
3 0.020 2 0.06 0.07 0.025833
4 0.010 3 0.04 0.08 0.027520
5 0.001 6 0.01 0.07 0.025833
從圖上和訊息中,我們可以看出error隨著cp的縮小,而遞減的速度。
~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~
上一篇: R上的CART Package — rpart [入門篇]
下一篇:
~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~